Инструмент оценки качества работы алгоритмов разметки медицинских изображений
Хакатон "Лидеры Цифровой Трансформации"
Приложение можно запустить через докер. Требуется установить docker.
Замените /home/glyc/Documents/banh_bao/data на абсолютный путь до папки data, который начинается c / или с ~/.
Тренировка модели занимает около 15 минут.
docker build -t train_image --target train .
docker run --rm -v /home/glyc/Documents/banh_bao/data:/data -v /home/glyc/Documents/banh_bao/data:/data --name bao_train train_image
Предсказания для тестового пака находятся по пути
./data/processed/test_predictions.csv
docker build -t predict_image --target predict .
docker run -d --rm -p 8501:8501 --name bao_predict predict_image
Откройте в браузере ссылку http://localhost:8501/.
Загрузите 3 изображения - оригинал, разметка эксперта и разметка модели - в соответствующие поля. Сервис покажет оценку разметки и shapley-значения.
Остановите сервис при помощи команды
docker stop bao_predict
docker build -t interactive_image --target interactive .
docker run -d --rm -p 8501:8501 --name bao_interactive interactive_image
Загрузите 2 изображения - оригинал и разметку эксперта - в соответствующие поля. Рисуйте свою разметку на изображении. Сервис покажет оценку Вашей разметки в реальном времени.
Откройте в браузере ссылку http://localhost:8501/.
Остановите сервис при помощи команды
docker stop bao_interactive
conda create --name banhbao
conda activate banhbao
pip install -r requirements/torch.txt
pip install -r requirements/train.txt
pip install -e .
cd streamlit
stramlit run evaluate.py
# open localhost:8501
Полученная модель позволяет сравнивать два снимка сегментационной разметки. Общая идеалогия такая - подобрать и придумать репрезентативные признаки. Обучить на этих признаках модель. Постпроцессинг предсказаний модели.
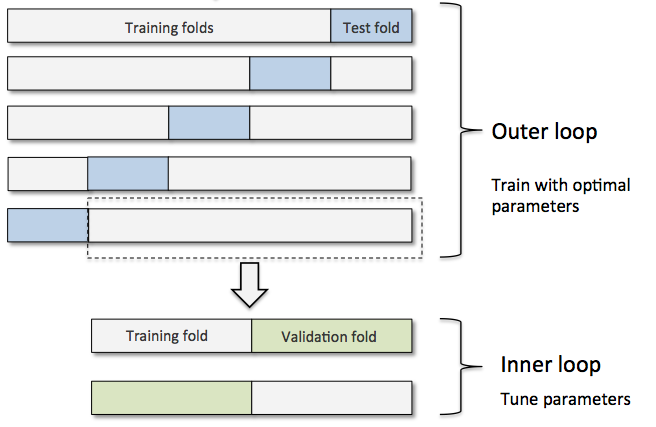
Для обучения модель использовался фреймворк lightGBM. Так как данных мало использовалась вложенная кросс-валидация.
Предположение - используем маски, которые врач оценил как 5 - то есть очень похожие на разметку врача. Выбранные маски можно принять экспертными и сравнить их с двумя другими. Оценки в этом случае не поменяются.
Добавлена аугментация меняющая тип разметки с овальной на квадратную и наоборот.
Описанные метрики находятся в metrics/run_metrics.py
IOU
DICE
F1 по объектам
Recall
Precision
Intersection over max - пересечение деленное на максимальную площадь объекта
Intersection over min - пересечение деленное на минимальную площадь объекта
Наличие найденных патологий - ввели бинарный признак показывающий - есть ли на маске патологии или нет.
Количество главных компонент каждой маски и их абсолютная разность.
Метрика - Structural Similarity Map взятая из статьи
Расстояние между поверхностями и среднее расстояние между поверхностями. Поверхность - это патолгоия на маске. репозиторий
Площади экспертной маски и маски модели, а также их абсолютная разность
Разные болезни могут иметь разный геометрический вид разметки. Также разные типы болезней может быть сложнее найти. Для предсказания типа болезни был использован репозиторий torchxrayvision, который дает предсказания болезней на 15 классов на датасете NIH.
Если разметка находится не в зоне легких, то это грубая ошибка. Для сегментации зоны с легкими исопльзовался lungs_finder. Использовали два режима - один выделяет кажду область легкого, второй, объединяет две области в одну, заполняя промежуток между ними и дополняя до прямоугольника. Если легкие не были найдены, то маской с легкими считается весь снимок. Для выделения областей не входящих в маску легких использовалась следущая последовательность побитовых операций:
diff_mask = XOR(lungs_mask, model_mask)
result = AND(diff_mask, model_mask)
Для нормализации метрики была посчитана площадь result и поделена на площадь первоначальной маски предсказания.
Был вычислен общий центр масс всех патологий на всей маске и центр масс маски сегментирующей легкие, спасибо lungs_finder. Метрика - положение центра масс патологий относительно центра масс легких.
Также сравнили патологии описанные своим центром масс попарно. Нашли пары ближайших друг к другу. И нашли среднее/max/min из всех расстояний в парах.
(c) Team "Бань Бао"