代码地址:https://github.com/RManLuo/neusomatic.git
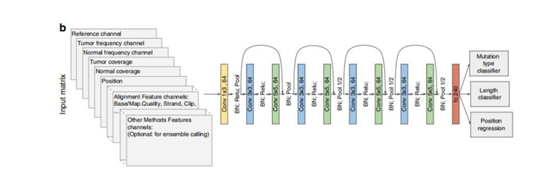
本文 提出了 NeuSomatic的方法 采用了深度学习对 体 细胞的 变异进行了异常检测 其表现显著优于之前的方法。使用 NeuSomatic,可以提高准确检查出靶点变异的几率,避免漏检从而更高效的指导他人精准用药。达到之前方法相同的准确度,需要的测序数据也会有显著的降低,从而可以降低检测的成本。 该方法的创新点在于将其他检测方法的输出和中间结果等作为输入, 从而做到了再一个框架下整合多种方法。 此方法 ,通过把序列 比对变成 小矩阵并且 使用了 100多个特征 ,达到有效的捕获突变信号 。
整体流程为:在预处理阶段(preprocess),将输入的tumor,normal,reference预处理整合成多个.tsv文件。 预测阶段(call),将输入的.tsv文件按照图中所示的resnet模型,输入进去。经过后处理阶段(postprocess),将预测阶段输出的文件转换成标准vcf格式。
- 实验采用 Homo_sapiens.GRCh37.75.dna.chromosome.22.fa.gz作为reference
- 肿瘤数据来源ICGC-TCGA-DREAM Somatic Mutation Calling Challenge ICGC-TCGA-DREAM Somatic Mutation Calling Challenge
- tumor.bam 作为肿瘤变异数据
- normal.bam 作为正常数据
- region.bed 作为整个训练的区域
BAM是SAM文件的二进制格式。 SAM(Sequence Alignment/Map)格式是一种通用的比对格式,用来存储reads到参考序列的比对信息。 SAM是一种序列比对格式标准,由sanger制定,是以TAB为分割符的文本格式。主要应用于测序序列mapping到基因组上的结果表示,当然也可以表示任意的多重比对结果。 SAM分为两部分,注释信息(header section)和比对结果部分(alignment section)。
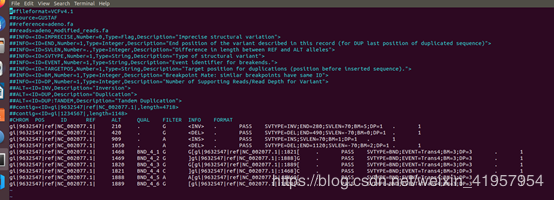
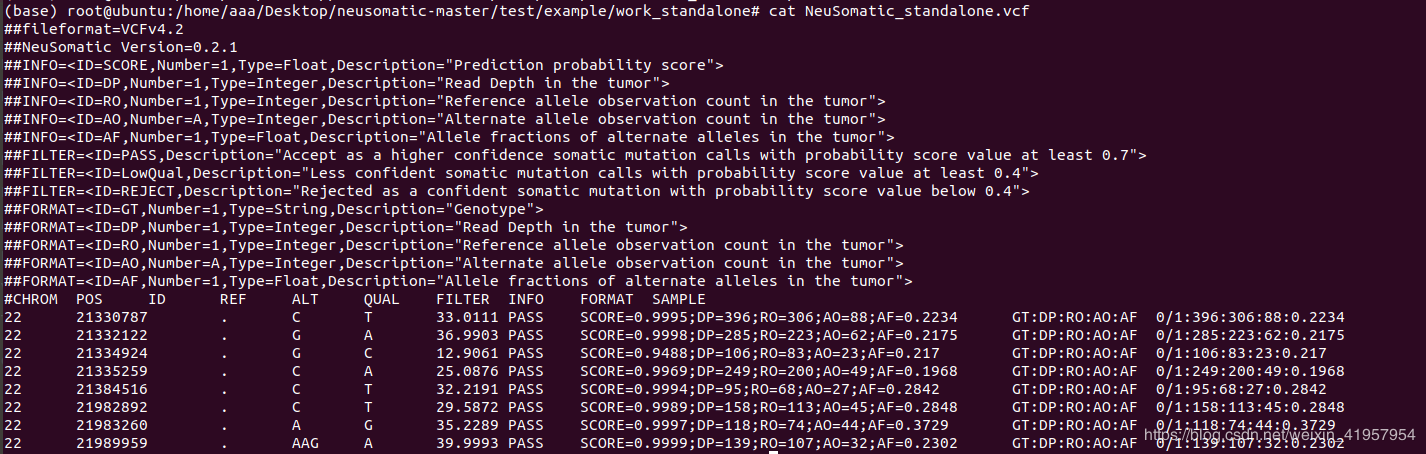
VCF是Variant Call Format的简称,是一种定义的专门用于存储基因序列突变信息的文本格式。在生物信息分析中会大量用到VCF格式。例如基因组中的单碱基突变,SNP, 插入/缺失INDEL, 拷贝数变异CNV,和结构变异SV等,都是利用VCF格式来存储的。将其存储为二进制格式就是BCF。
- CHROM [chromosome]: 染色体名称,
- POS [position]: 参考基因组突变碱基位置,如果是INDEL,位置是INDEL的第一个碱基位置。
- ID [identifier]: 突变的名称,
- REF [reference base(s)]:参考染色体的碱基
- ALT [alternate base(s)]: 与参考序列比较,发生突变的碱基,
- QUAL [quality]: Phred标准下的质量值
- FILTER [filter status]:使用其它的方法进行过滤后得到的过滤结果
- INFO
FASTA文件主要用于存储生物的序列文件,例如基因组,基因的核酸序列以及氨基酸等,是最常见的生物序列格式,一般以扩展名fa,fasta,fna等。fasta文件中,第一行是由大于号">"开头的任意文字说明,用于序列标记,为了保证后续分析软件能够区分每条序列,单个序列的标识必须是唯一的,序列ID部分可以包含注释信息。从第二行开始为序列本身,只允许使用既定的核苷酸或氨基酸编码符号。序列部分可以在一行,也可以分成多行。
BED 文件格式提供了一种灵活的方式来定义的数据行,用于描述注释的信息。 跟GTF/GFF格式一样,也可以用来描述基因组特征。但没有GTF/GFF格式那么正规,通常用来描述 任何人为定义的区间。 但没有GTF/GFF格式那么正规,通常用来描述任何人为定义的区间。 所以BED格式最重要的就是染色体加上起始终止坐标这3列。
残差模块由:输入卷积层+BN层+卷积层+BN层+残差层+池化层组成。
class NSBlock(nn.Module):
def __init__(self, dim, ks_1=3, ks_2=3, dl_1=1, dl_2=1, mp_ks=3, mp_st=1):
super(NSBlock, self).__init__()
self.dim = dim
self.conv_r1 = nn.Conv2d(
dim, dim, kernel_size=ks_1, dilation=dl_1, padding=(dl_1 * (ks_1 - 1)) // 2)
self.bn_r1 = nn.BatchNorm2d(dim)
self.conv_r2 = nn.Conv2d(
dim, dim, kernel_size=ks_2, dilation=dl_2, padding=(dl_2 * (ks_2 - 1)) // 2)
self.bn_r2 = nn.BatchNorm2d(dim)
self.pool_r2 = nn.MaxPool2d((1, mp_ks), padding=(
0, (mp_ks - 1) // 2), stride=(1, mp_st))
def forward(self, x):
y1 = (F.relu(self.bn_r1(self.conv_r1(x))))
y2 = (self.bn_r2(self.conv_r2(y1)))
y3 = x + y2
z = self.pool_r2(y3)
return z网络结构:类似与Res-Net的残差结构,4个残差卷积block,每个block包含两个卷积层,一个BatchNormalize层,一个池化层,开始有个1X3的卷积层,最后两层FC层,在输出层,作者用了两个softma层来输:变异类型:(non-somatic call, SNV, insertion, deletion)和变异长度:(0,1,2,>2);一个回归层来确定变异位点位置(1-32)。
class NeuSomaticNet(nn.Module):
def __init__(self, num_channels):
super(NeuSomaticNet, self).__init__()
dim = 64
self.conv1 = nn.Conv2d(num_channels, dim, kernel_size=(
1, 3), padding=(0, 1), stride=1)
self.bn1 = nn.BatchNorm2d(dim)
self.pool1 = nn.MaxPool2d((1, 3), padding=(0, 1), stride=(1, 1))
self.nsblocks = [
[3, 5, 1, 1, 3, 1],
[3, 5, 1, 1, 3, 2],
[3, 5, 2, 1, 3, 2],
[3, 5, 4, 2, 3, 2],
]
res_layers = []
for ks_1, ks_2, dl_1, dl_2, mp_ks, mp_st in self.nsblocks:
rb = NSBlock(dim, ks_1, ks_2, dl_1, dl_2, mp_ks, mp_st)
res_layers.append(rb)
self.res_layers = nn.Sequential(*res_layers)
ds = np.prod(list(map(lambda x: x[5], self.nsblocks)))
self.fc_dim = dim * 32 * 5 // ds
self.fc1 = nn.Linear(self.fc_dim, 240)
self.fc2 = nn.Linear(240, 4)
self.fc3 = nn.Linear(240, 1)
self.fc4 = nn.Linear(240, 4)
def forward(self, x):
x = self.pool1(F.relu(self.bn1(self.conv1(x))))
internal_outs = [x]
x = self.res_layers(x)
internal_outs.append(x)
x2 = x.view(-1, self.fc_dim)
x3 = F.relu(self.fc1(x2))
internal_outs.extend([x2, x3])
o1 = self.fc2(x3)
o2 = self.fc3(x3)
o3 = self.fc4(x3)
return [o1, o2, o3], internal_outs- 执行
.build.sh生成scan_alignments的二进制文件,该文件可以用来生成对其的序列。 - 预处理部分对应于项目中的preprocessing模块。执行以下代码,可以把输入的
turmor.bam,normal.bam和region.bed结合训练数据
python preprocess.py \
--mode train \
--reference GRCh38.fa \
--region_bed region.bed \
--tumor_bam tumor.bam \
--normal_bam normal.bam \
--work work_train \
--truth_vcf truth.vcf \
--min_mapq 10 \
--number_threads 10 \
--scan_alignments_binary ../bin/scan_alignments
Mode 参数候选项有train 以及call 两种模式。
Reference 参数对应的为 输入中的 reference channel
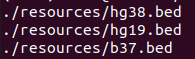
Region_bed 项目中除了default的以外还提供了
Tumor.bam以及normal.bam 对应的输入 tumor channel,normal channel
- 训练神经网络模块:
python train.py \
--candidates_tsv work_train/dataset/*/candidates*.tsv \
--out work_train \
--num_threads 10 \
--batch_size 100
--candidates_tsv 即对应 preprocessing 的输出。 --reference 同上 --out 目标文件夹 --checkpoint 直接调用以及训练好的参数模型
- 后处理阶段,把训练阶段的预测结果转换成标准的vcf格式
python postprocess.py \
--reference GRCh38.fa \
--tumor_bam tumor.bam \
--pred_vcf work_call/pred.vcf \
--candidates_vcf work_call/work_tumor/filtered_candidates.vcf \
--output_vcf work_call/NeuSomatic.vcf \
--work work_call
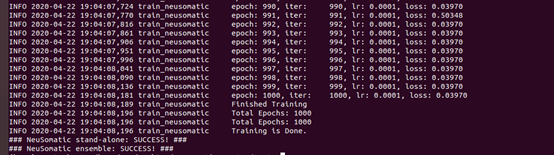
训练bash:
#!/bin/bash
set -e
test_dir="$( cd "$( dirname "${BASH_SOURCE[0]}" )" >/dev/null && pwd )"
neusomatic_dir="$( dirname ${test_dir} )"
cd ${test_dir}
mkdir -p example
cd example
if [ ! -f Homo_sapiens.GRCh37.75.dna.chromosome.22.fa ]
then
if [ ! -f Homo_sapiens.GRCh37.75.dna.chromosome.22.fa.gz ]
then
wget ftp://ftp.ensembl.org/pub/release-75//fasta/homo_sapiens/dna/Homo_sapiens.GRCh37.75.dna.chromosome.22.fa.gz
fi
gunzip -f Homo_sapiens.GRCh37.75.dna.chromosome.22.fa.gz
fi
if [ ! -f Homo_sapiens.GRCh37.75.dna.chromosome.22.fa.fai ]
then
samtools faidx Homo_sapiens.GRCh37.75.dna.chromosome.22.fa
fi
#Ensemble NeuSomatic train
python ${neusomatic_dir}/neusomatic/python/preprocess.py \
--mode train \
--reference Homo_sapiens.GRCh37.75.dna.chromosome.22.fa \
--region_bed ${test_dir}/region.bed \
--tumor_bam ${test_dir}/tumor.bam \
--normal_bam ${test_dir}/normal.bam \
--work work_train \
--min_mapq 10 \
--num_threads 1 \
--truth_vcf ${test_dir}/train.vcf \
--scan_alignments_binary ${neusomatic_dir}/neusomatic/bin/scan_alignments
CUDA_VISIBLE_DEVICES= python ${neusomatic_dir}/neusomatic/python/train.py \
--candidates_tsv work_train/dataset/*/candidates*.tsv \
--out work_train \
--num_threads 1 \
--batch_size 100
cd ..
file1=${test_dir}/example/work_standalone/NeuSomatic_standalone.vcf
file2=${test_dir}/NeuSomatic_standalone.vcf
cmp --silent $file1 $file2 && echo "### NeuSomatic stand-alone: SUCCESS! ###" \
call_bash:
#!/bin/bash
set -e
test_dir="$( cd "$( dirname "${BASH_SOURCE[0]}" )" >/dev/null && pwd )"
neusomatic_dir="$( dirname ${test_dir} )"
cd ${test_dir}
mkdir -p example
cd example
if [ ! -f Homo_sapiens.GRCh37.75.dna.chromosome.22.fa ]
then
if [ ! -f Homo_sapiens.GRCh37.75.dna.chromosome.22.fa.gz ]
then
wget ftp://ftp.ensembl.org/pub/release-75//fasta/homo_sapiens/dna/Homo_sapiens.GRCh37.75.dna.chromosome.22.fa.gz
fi
gunzip -f Homo_sapiens.GRCh37.75.dna.chromosome.22.fa.gz
fi
if [ ! -f Homo_sapiens.GRCh37.75.dna.chromosome.22.fa.fai ]
then
samtools faidx Homo_sapiens.GRCh37.75.dna.chromosome.22.fa
fi
rm -rf work_ensemble
#Ensemble NeuSomatic test
python ${neusomatic_dir}/neusomatic/python/preprocess.py \
--mode call \
--reference Homo_sapiens.GRCh37.75.dna.chromosome.22.fa \
--region_bed ${test_dir}/region.bed \
--tumor_bam ${test_dir}/tumor.bam \
--normal_bam ${test_dir}/normal.bam \
--work work_ensemble \
--scan_maf 0.05 \
--min_mapq 10 \
--snp_min_af 0.05 \
--snp_min_bq 20 \
--snp_min_ao 10 \
--ins_min_af 0.05 \
--del_min_af 0.05 \
--num_threads 1 \
--ensemble_tsv ${test_dir}/ensemble.tsv \
--scan_alignments_binary ${neusomatic_dir}/neusomatic/bin/scan_alignments
CUDA_VISIBLE_DEVICES= python ${neusomatic_dir}/neusomatic/python/call.py \
--candidates_tsv work_ensemble/dataset/*/candidates*.tsv \
--reference Homo_sapiens.GRCh37.75.dna.chromosome.22.fa \
--out work_ensemble \
--checkpoint ${neusomatic_dir}/neusomatic/models/NeuSomatic_v0.1.0_ensemble_Dream3_70purity.pth \
--num_threads 1 \
--ensemble \
--batch_size 100
python ${neusomatic_dir}/neusomatic/python/postprocess.py \
--reference Homo_sapiens.GRCh37.75.dna.chromosome.22.fa \
--tumor_bam ${test_dir}/tumor.bam \
--pred_vcf work_ensemble/pred.vcf \
--candidates_vcf work_ensemble/work_tumor/filtered_candidates.vcf \
--ensemble_tsv ${test_dir}/ensemble.tsv \
--output_vcf work_ensemble/NeuSomatic_ensemble.vcf \
--work work_ensemble
cd ..
file1=${test_dir}/example/work_standalone/NeuSomatic_standalone.vcf
file2=${test_dir}/NeuSomatic_standalone.vcf
cmp --silent $file1 $file2 && echo "### NeuSomatic stand-alone: SUCCESS! ###" \
|| echo "### NeuSomatic stand-alone FAILED: Files ${file1} and ${file2} Are Different! ###"
file1=${test_dir}/example/work_ensemble/NeuSomatic_ensemble.vcf
file2=${test_dir}/NeuSomatic_ensemble.vcf
cmp --silent $file1 $file2 && echo "### NeuSomatic ensemble: SUCCESS! ###" \
|| echo "### NeuSomatic ensemble FAILED: Files ${file1} and ${file2} Are Different! ###"
实验结果:
对应vcf的字段可以在上文vcf文件格式出参考。可以预测出每条序列的变异类型、 变异长度和变异位点位置。